La scoperta viene da un gruppo di ricercatori del Centro della Complessità e dei Biosistemi dell’Università Statale di Milano che ha identificato un “trait d’union” tra le tre patologie: 38 geni associati all’infiammazione in corso e alla risposta immunitaria.
Epidemiologia e clinica le associano già da tempo
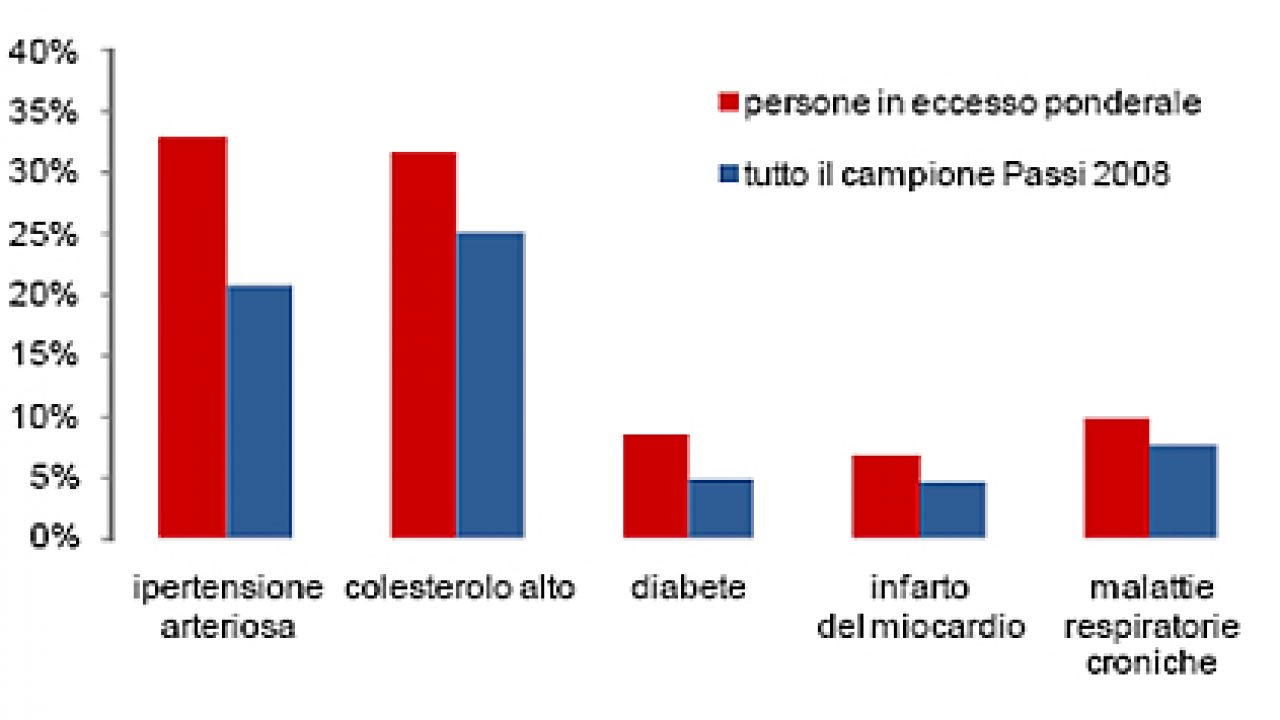
Sono molti i dati epidemiologici e gli studi clinici che documentano l’associazione tra obesità, in continua crescita, è diabete di tipo 2: si parla di diabesità (neologismo coniato dall’Organizzazione Mondiale della Sanità) in oltre il 70% dei casi, con punte dell’80-90%. L’obesità si associa anche a diversi tipi di tumore, tra questi, nelle donne, il carcinoma della mammella. Nonostante la vasta letteratura che suggeriva la triplice associazione, fino ad oggi non era ancora stato trovato un legame genetico che le collegasse.
38 geni dall’espressione alterata: la “firma” che accomuna le tre patologie
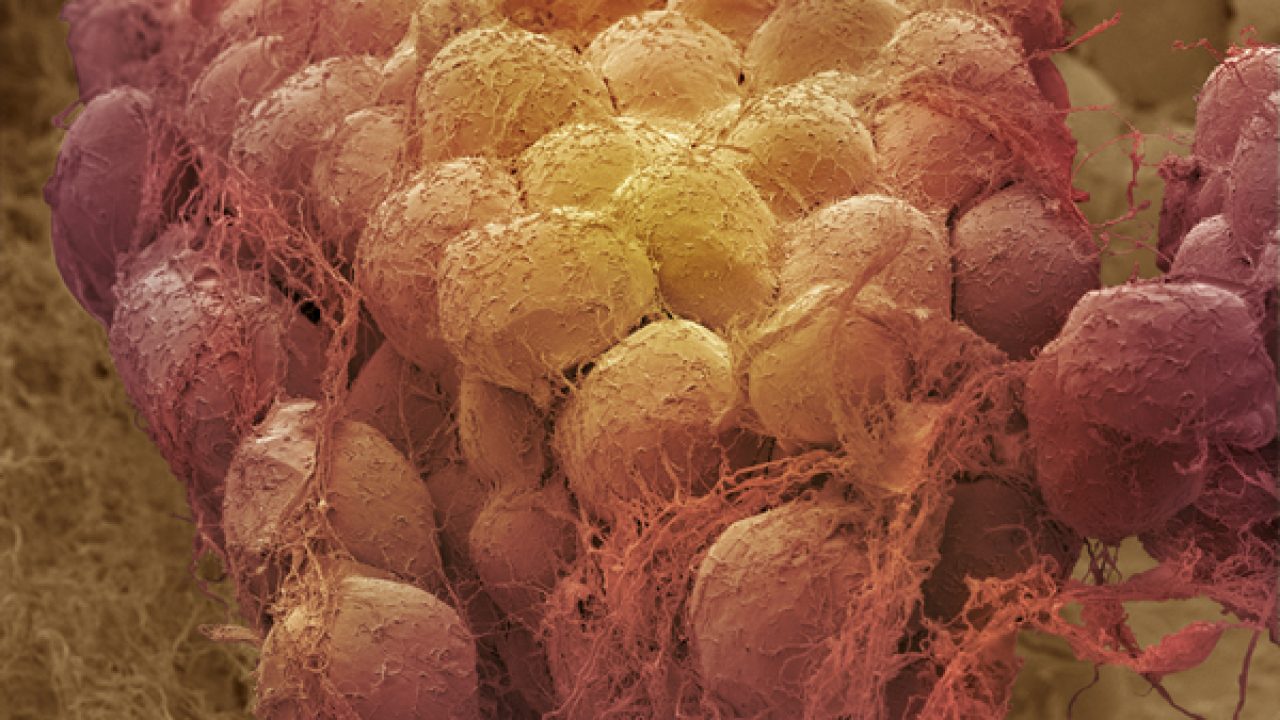
I ricercatori italiani hanno individuato 38 geni che negli adipociti (le cellule del tessuto adiposo, dove si accumula il grasso) si esprimono in maniera diversa nelle persone con obesità rispetto a quelle in normopeso. Gli studiosi hanno notato che si tratta di una serie di geni correlati soprattutto a processi infiammatori, risposta immunitaria e complicanze tipiche dell’obesità come il diabete di tipo 2, ma non solo. Hanno riscontrato alterazioni genetiche simili anche nelle donne con un cancro alla mammella. Tale scoperta, oltre ad essere significativa dal punto di vista scientifico, potrebbe avere delle ricadute pratiche: secondo gli studiosi, infatti, alcuni dei 38 geni isolati, potrebbero venire usati come nuovi marcatori tumorali, un utilizzo importante sia per la ricerca scientifica sia per la diagnostica clinica.
Un approccio innovativo ai big data per isolare i geni coinvolti
Riuscire a identificare con precisione i 38 geni non è stato per nulla facile, hanno commentato i ricercatori. Il problema principale era isolare i dati ricorrenti di interesse nell’enorme massa di dati raccolti da diversi database che costituiva un assordante “rumore di fondo”, un effetto tecnico che gli esperti chiamano “batch effect”, effetto che può mascherare alcune differenze importanti dal punto di vista biologico quando si confrontano campioni che provengono da set di dati differenti. Come sono riusciti a superare il problema? I ricercatori hanno utilizzato un approccio innovativo – combinando due diverse tecniche informatiche – che ha consentito di mitigare il “rumore di fondo”. Questa strategia di analisi potrebbe avere ulteriori utilizzi in futuro, consentendo di studiare altre patologie con maggiore accuratezza, grazie all’enorme mole di dati raccolti dalla letteratura biomedicale – ha spiegato la d.ssa Caterina La Porta, membro del CC&B e docente di patologia generale al Dipartimento di Scienze e Politiche ambientali dell’Università di Milano. “Grazie a questo approccio, siamo riusciti a identificare una lista di geni caratteristici dell’obesità, che sono anche associati al diabete di tipo 2 e al cancro alla mammella. Il tutto con un grado di precisione simile a quello usato per identificare il Bosone di Higgs”, paragone suggestivo con la fisica delle particelle a indicare che l’approccio utilizzato consente un notevole grado di accuratezza dell’analisi.
Fonti
– Font-Clos F, Zapperi S, La Porta CAM – Integrative analysis of pathway deregulation in obesity. NPJ Syst Biol Appl 2017 Jun 30;3:18
– Centro della Complessità e dei Biosistemi, CC&B, Università di Milano